Osservabilità di un link e come valutarla
Abbiamo proposto un nuovo metodo per identificare la connettività di una rete basato sull'analisi nel dominio dei tempi del coefficiente di cross-correlazione di Pearson valutato a ritardo nullo e della sua significatività. Un obiettivo principale è quello di comprendere a quale scala tempi la connettività si manifesta. Per stimare il livello di significatività dei coefficienti di correlazione sperimentali e dunque il livello di connettività, è stata presa in considerazione un'ipotesi realistica e stringente, valutata generando serie temporali surrogate. Utilizzando dati MEG pubblicamente disponibili di soggetti sani in resting-state, e investigando la cross-correlazione tra nodi a scale tempi da 0.5 s a 60 s, abbiamo individuato strutture a rete simili a quelle descritte nella letteratura scientifica, purché la finestra di correlazione sia scelta sufficientemente ampia. Il metodo è descritto in:
A. Perinelli, D. E. Chiari and L. Ricci, Correlation in brain networks at different time scale resolution, Chaos 28 (2018) p. 063127 doi:10.1063/1.5025242
Il metodo è anche disponibile come pacchetto software open-source, il pacchetto NetOnZeroDXC (vai alla repository GitHub). Il pacchetto è descritto nel dettaglio in:
A. Perinelli, L. Ricci, NetOnZeroDXC: A package for the identification of networks out of multivariate time series via zero-delay cross-correlation, SoftwareX 10 (2019), 100316, doi:10.1016/j.softx.2019.100316

I link osservabili tra aree del cervello sono dovuti alla vicinanza spaziale?
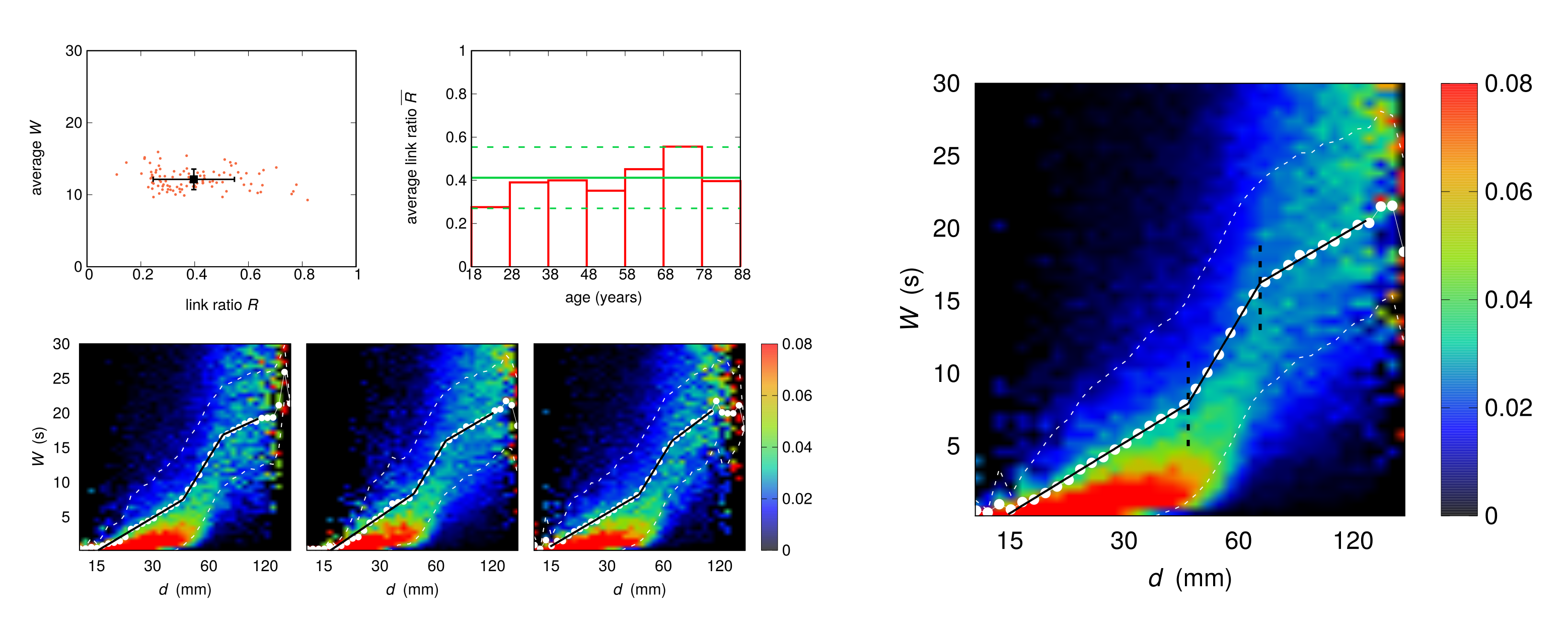
In ogni rete, la dipendenza della connettività dalla distanza fisica tra i nodi è una diretta conseguenza di compromessi tra i costi di creazione e mantenimento di un link, la velocità di propagazione dei segnali tra i nodi, e la capacità computazionale della rete. Nonostante l'universalità di questo problema, esso è affrontato in un numero ancora scarso di studi. Abbiamo applicato il nostro metodo di cross-correlazione a registrazioni MEG di resting-state e abbiamo analizzato la forza della connettività in funzione della distanza fisica tra i nodi. La dipendenza tra scala tempi di osservabilità e lunghezza di un link è descrivibile da una legge a potenza caratterizzata da un esponente la cui entità è inversamente proporzionale alla connettività. Questi risultati sono pubblicati in:
A. Perinelli, D. Tabarelli, C. Miniussi and L. Ricci, Dependence of connectivity on geometric distance in brain networks, Scientific Reports 9 (2019), 13412, doi:10.1038/s41598-019-50106-2
Ulteriori indagini sulla relazione tra connettività e distanza
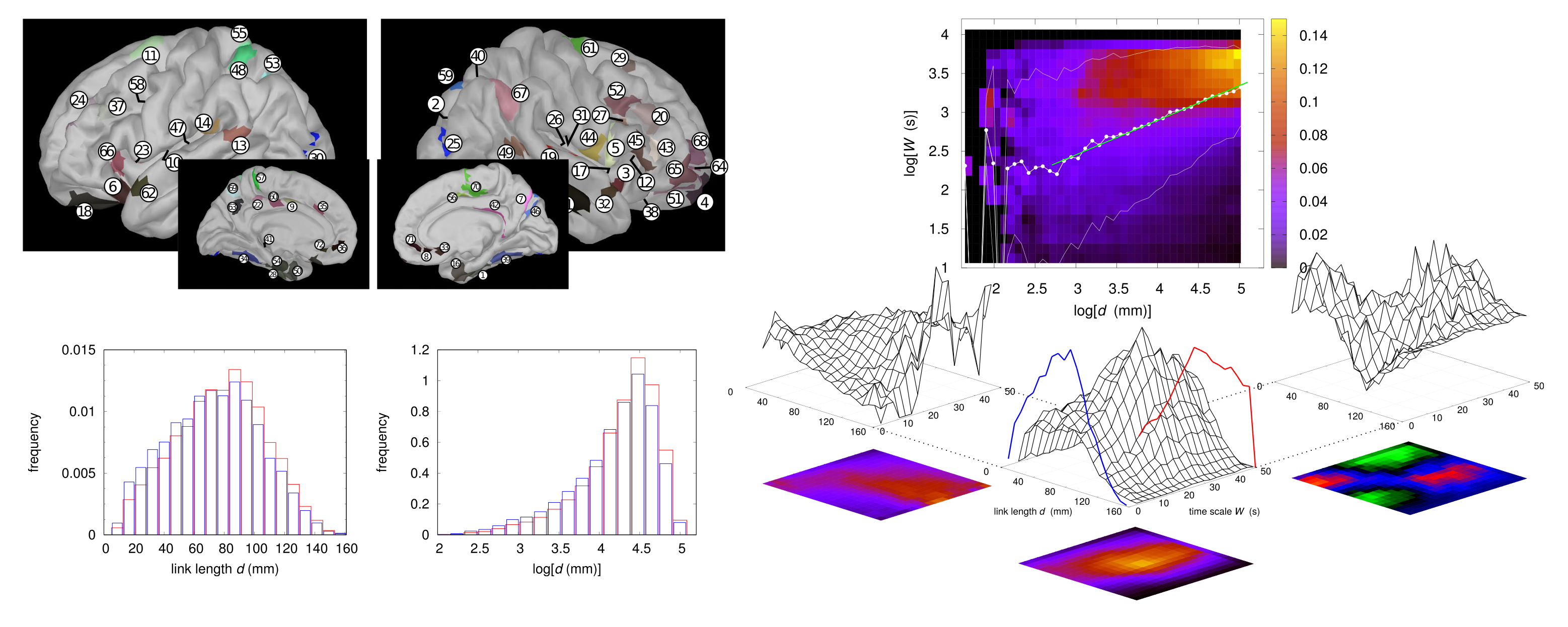
L'analisi precedente è stata migliorata utilizzando un dataset più grande contenente dati MEG di 100 individui sani in resting-state e, inoltre, aggiornando gli strumenti di analisi. In aggiunta, coppie di nodi sono state distinte tra inter-emisferiche e intra-emisferiche per evidenziare una possibile differenza in connettività tra la comunicazione all'interno o tra due diversi emisferi. Abbiamo ottenuto un'evidenza statisticamente rilevante di tre regimi di dipendenza lineare del tempo scala di osservabilità dal logaritmo naturale della distanza. Questo risultato è un interessante punto di partenza per investigazioni successive mirate, ad esempio, a relazionare i tra diversi regimi al principio di segregazione/integrazione supportato dalla topologia small-world delle reti. Questi risultati sono stati pubblicati in:
M. Castelluzzo, A. Perinelli, D. Tabarelli and L. Ricci, Dependence of connectivity on the logarithm of geometric distance in brain networks, Frontiers in Physiology 11 (2021), 611125, doi:10.3389/fphys.2020.611125